Genome-In-A-Bottle (GIAB; versión 3.3.2) contiene 3,21 M SNP en cromosomas auto + X y 0,51 M INDEL en regiones de confianza de 2,58 Gb. La relación ins: del es 0.92. En el conjunto pacbio CHM1-CHM13 (ascendencia europea), hay 3,57 M auto + X SNP y 0,58 M INDEL en regiones seguras de 2,71 Gb [ referencia] con una relación ins: del 0,99. Estos le dan una idea de los INDEL relativamente cortos.
Para indels de más de 50 pb, le recomiendo leer este artículo de Chaisson et al. Este conjunto de llamadas se construye a partir del ensamblaje del genoma completo del genoma CHM1. Es mucho más completo y probablemente más preciso que todos los demás conjuntos de llamadas. La siguiente es la Tabla 1 del artículo:
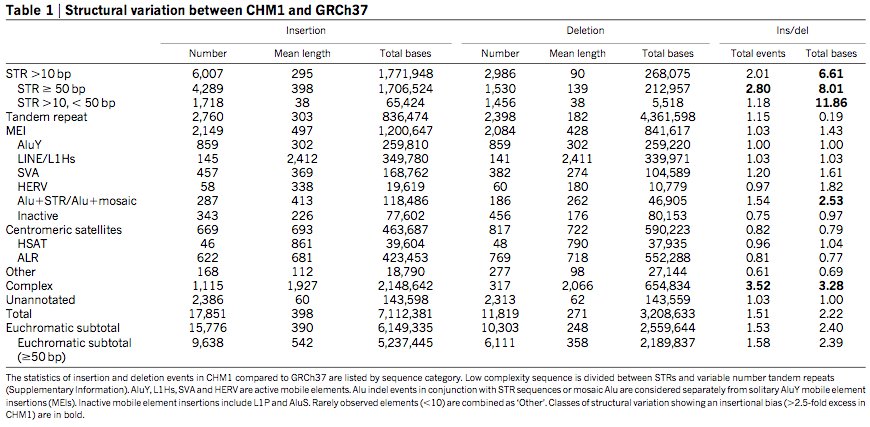
Tengo que decir que la alta proporción de ins-/ del-del-ratio me sorprende , pero esto es lo que nos dicen los datos. Hice un análisis similar y alcancé una proporción similar.
PD: Estos números son esencialmente de un haplotipo. Serán más altos para una muestra diploide.
EDITAR: algunos números en la publicación original son incorrectos.